Chapter 5 生存曲線
5.1 生存時間解析
時間の経過の中で、あることが{起こる・起こらない}ということを調べるために、カプラン・マイヤー曲線を用います。 この時、黄砂がある場合には
- カプラン・マイヤー曲線の作図
- 検定
- log-rank 検定 (または一般化 Wilcoxon 検定)
- Cox 比例ハザードモデル
検定は、2群の比較しかできません。また、カプラン・マイヤー曲線が交差する場合は、検定する前に原因を見つける必要があります。
5.2 論文 Dessu (2020) PLoS One
Dessu S, Habte A, & Mesele M (2020). The Kaplan Meier estimates of mortality and its predictors among newborns admitted with low birth weight at public hospitals in Ethiopia. PloS one, 15(9), e0238629. https://doi.org/10.1371/journal.pone.0238629
新生児のうち、2500グラム未満で生まれる事例が全世界で毎年2000万件を超えています。2500グラム未満では免疫系が十分に発達しておらず、死亡リスクが高い状態にあります。
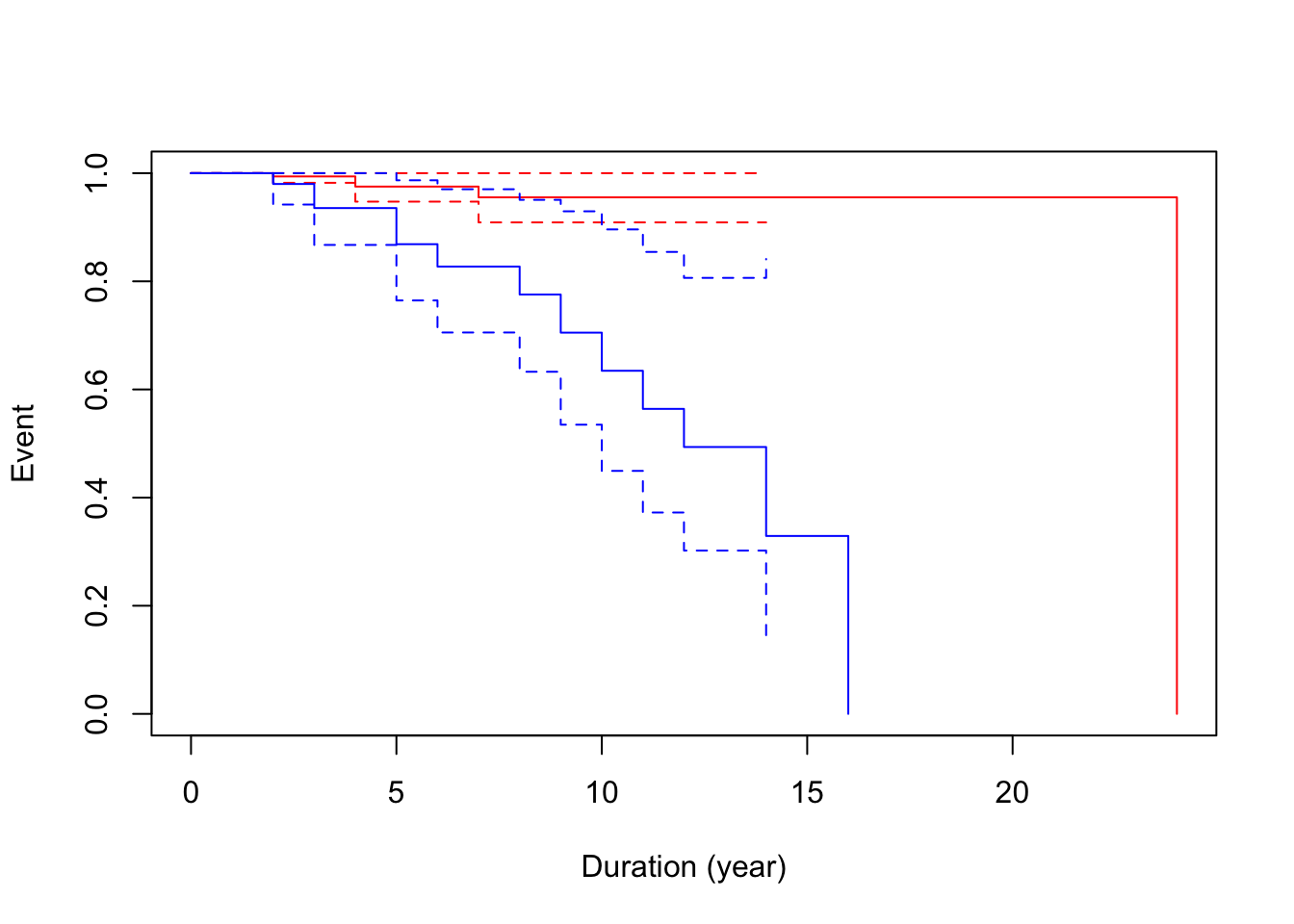
カプランマイヤー図は3つあります。
- Fig 1:エピオピアの公立病院における2500グラム未満児の生存曲線
- Fig 2:1時間以内に Kangaroo Mother Care (KMC) を開始した群とそうでない群
- Fig 3:1時間以内に exclusive breast feeding を開始した群とそうでない群
ここでは、Fig 2を再現してみましょう。
knitr::include_graphics("img/pone.0238629.g002.PNG_L.png")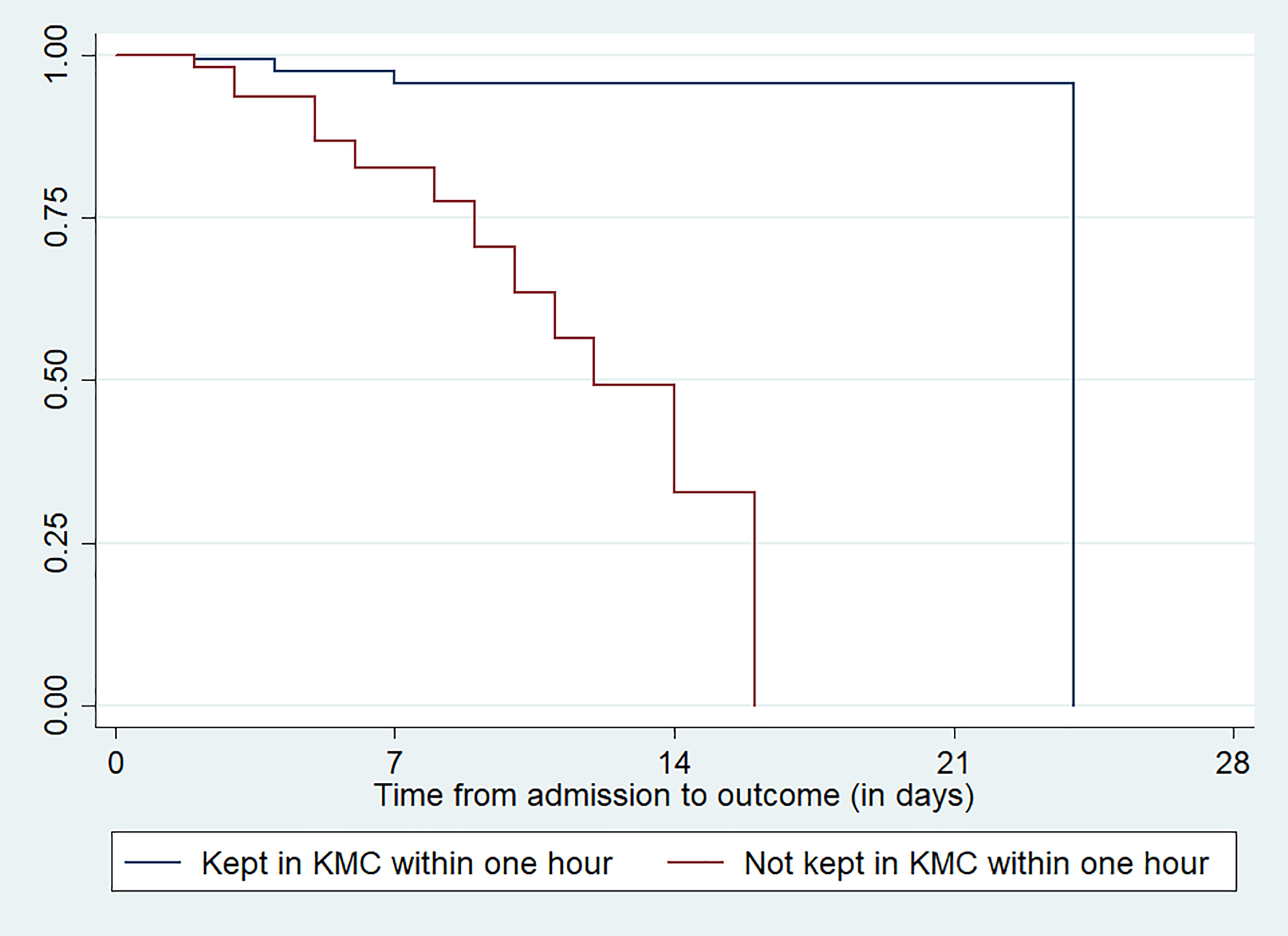
Figure 5.1: Dessu (2020) Fig 2. Survival estimation of neonates admitted with low birth weight for the variable kept the neonates in KMC within one hour at public hospitals in Ethiopia
5.3 STATA データの読み込み
データを読み込みます。
library(haven)
dfSurvival <- read_stata("https://doi.org/10.1371/journal.pone.0238629.s001")
dfSurvival$KMC <- as.factor(dfSurvival$KMC)生存分析の統計に先立ち、オブジェクトを準備します。
library(survival)
objSurv <- survfit(formula = Surv(stime,Died) ~ KMC,
data = dfSurvival)
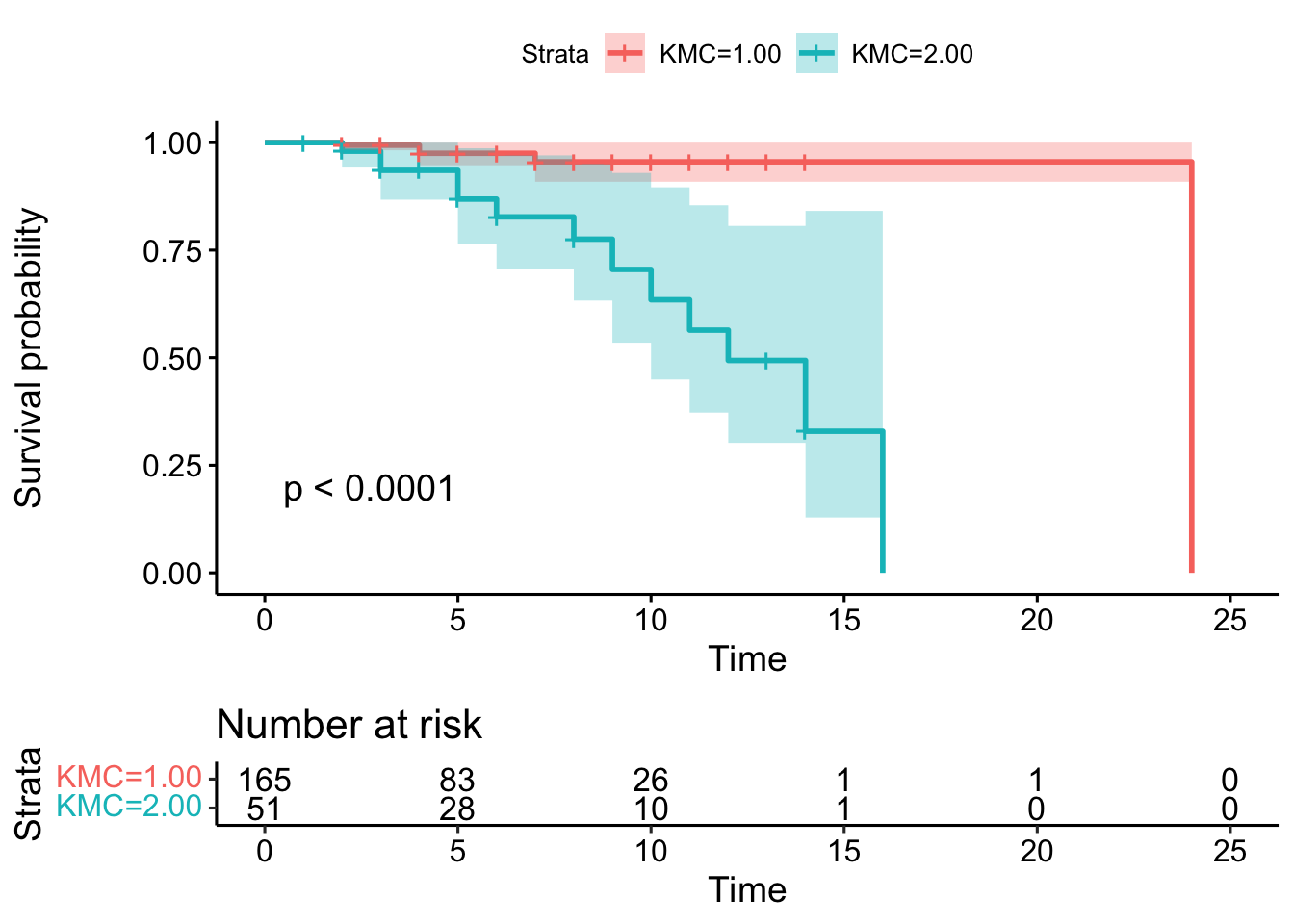
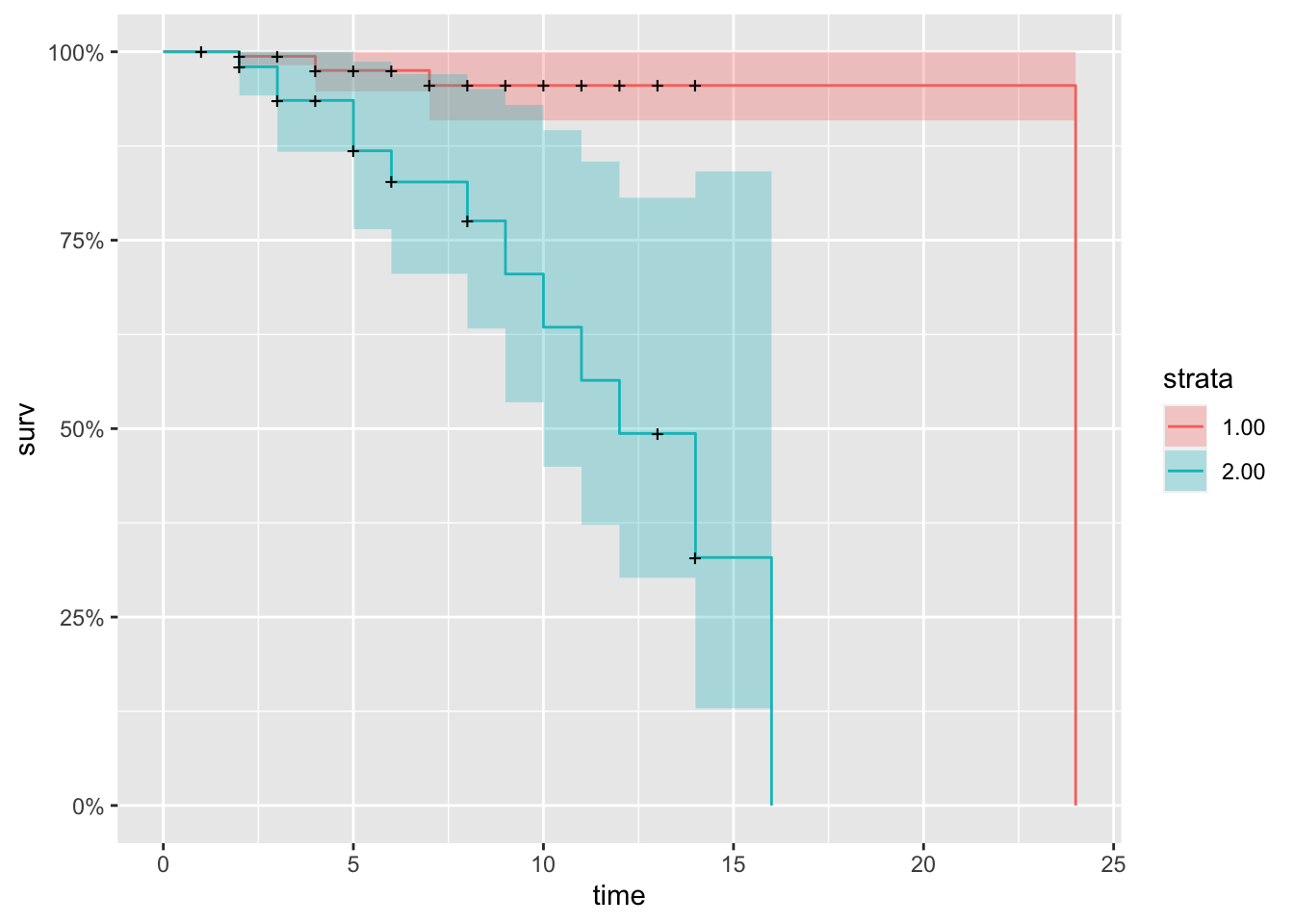
summary(objSurv)## Call: survfit(formula = Surv(stime, Died) ~ KMC, data = dfSurvival)
##
## KMC=1.00
## time n.risk n.event survival std.err lower 95% CI upper 95% CI
## 2 165 1 0.994 0.00604 0.982 1
## 4 106 2 0.975 0.01441 0.947 1
## 7 49 1 0.955 0.02423 0.909 1
## 24 1 1 0.000 NaN NA NA
##
## KMC=2.00
## time n.risk n.event survival std.err lower 95% CI upper 95% CI
## 2 50 1 0.980 0.0198 0.942 1.000
## 3 44 2 0.935 0.0361 0.867 1.000
## 5 28 2 0.869 0.0565 0.765 0.987
## 6 21 1 0.827 0.0673 0.705 0.970
## 8 16 1 0.776 0.0805 0.633 0.951
## 9 11 1 0.705 0.0994 0.535 0.929
## 10 10 1 0.635 0.1117 0.449 0.896
## 11 9 1 0.564 0.1195 0.372 0.854
## 12 8 1 0.494 0.1236 0.302 0.806
## 14 3 1 0.329 0.1576 0.129 0.841
## 16 1 1 0.000 NaN NA NA変数
- objSurv: survival パッケージのオブジェクト。
- dfSurvival: 上で作ったデータフレーム。
関数
- survfit: survival パッケージの関数。生存時間データのカプランマイヤー推定で用いる関数
- Surv: survival パッケージの関数。生存時間データオブジェクトを作成する。
- summary: base 関数。
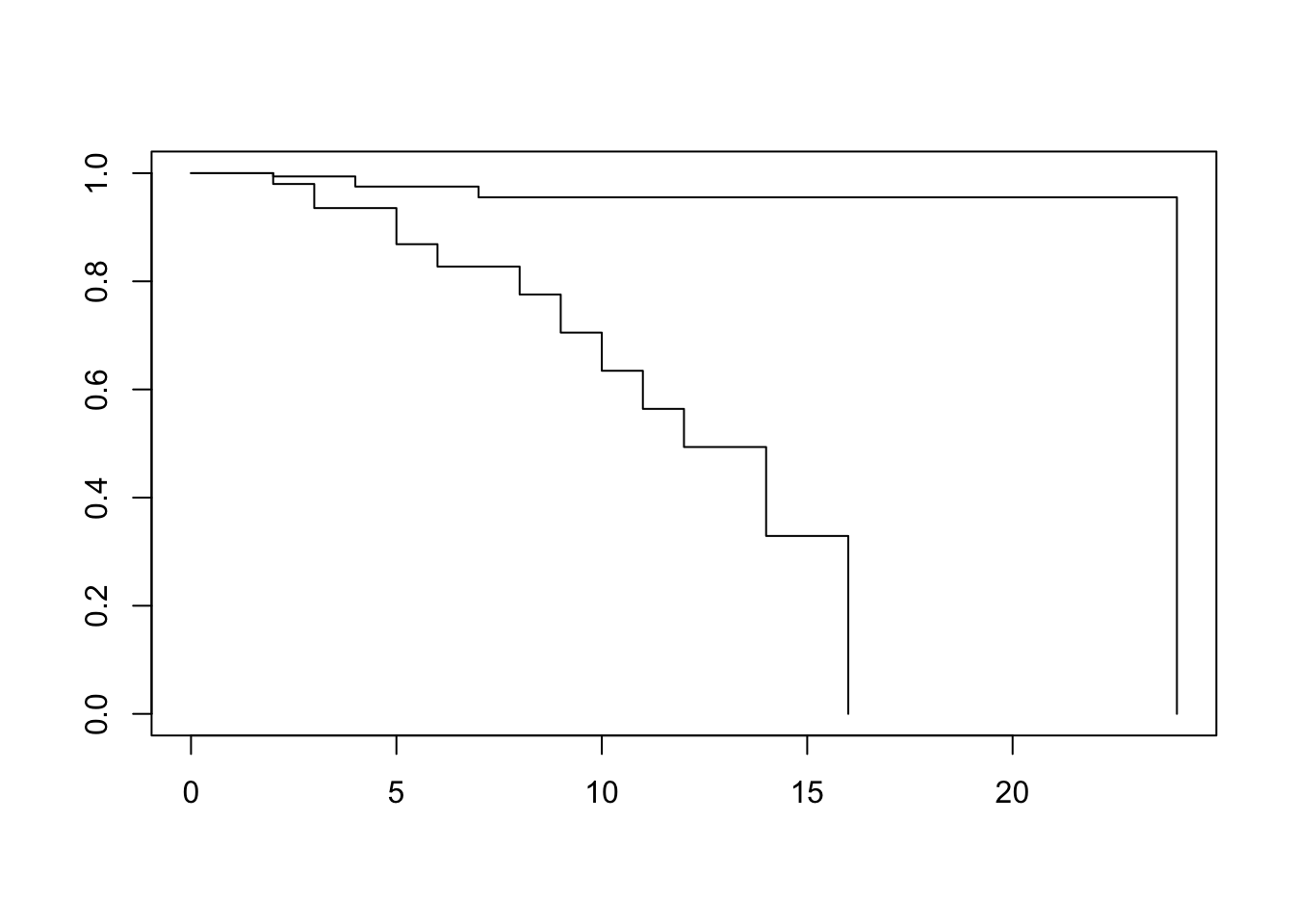
5.4 生存曲線の作成
生存曲線 (Kaplan-Meier 曲線) を描いてみましょう。
R で作図する場合、大きく分けて base 関数の plot を用いる方法と、多機能な ggplot2 ライブラリを用いる方法があります。
5.5 log-rank 検定
5.5.1 2群の比較 log-rank 検定
ログ・ランク (log-rank)検定は、群ごとのイベントありとなしに集計したクロス表のカイ2乗検定です。R の survival パッケージが自動的に計算します。
survdiff(Surv(stime,Died)~KMC,
data = dfSurvival) ## Call:
## survdiff(formula = Surv(stime, Died) ~ KMC, data = dfSurvival)
##
## N Observed Expected (O-E)^2/E (O-E)^2/V
## KMC=1.00 165 5 13.29 5.17 20.6
## KMC=2.00 51 13 4.71 14.57 20.6
##
## Chisq= 20.6 on 1 degrees of freedom, p= 6e-06p < 0.01 なので、この場合は群間差があったと判断します。
5.6 サンプルサイズ
library(powerSurvEpi)
library(npsurvSS)
library(gsDesign)5.6.2 Cox 比例ハザードモデル
- powerSurvEpi
- npsurvSS
- gsDesign
ライブラリ powerSurvEpi は、パイロット試験データを元に、Cox 比例ハザードモデルのサンプルサイズ計算を行うことができます。
library(powerSurvEpi)
dfSurvival$KMC2 <- factor(x = dfSurvival$KMC, levels = c("1.00", "2.00"), labels = c("C", "E"))
samplesize <- ssizeCT(formula = Surv(stime,Died) ~ KMC2,
dat = dfSurvival,
power = 0.80,
k = 1,
RR = 1.2,
alpha = 0.05)関数
- factor 因子型の列を作成します。元となる列はKMCで、値 (“1.00”, “2.00”) を (“C”, “E”) に割り当てています。
| 引数 | 内容 |
|---|---|
| formula | これまでも使って来た式です。目的変数は Surv 関数を使う必要があります。説明変数は、“C” と “E” からなる因子型である必要があります。 |
| k | 暴露群:対照群の比率 (k:1) です。 |
| RR | ハザード比の仮定値です。 |